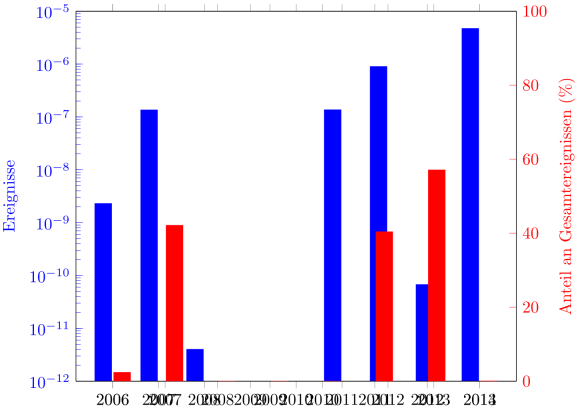
|
Ich denke, mein Problem wird mit folgendem Code deutlich: \documentclass{standalone} \usepackage{pgfplots} \pgfplotsset{compat=newest} \begin{document} \begin{tikzpicture} \begin{semilogyaxis}[ ybar, log origin=infty, width = 0.9\linewidth, ymin=1.0e-12, ymax=1.0e-05, ylabel={Ereignisse}, yticklabel style=blue, ytick style=blue, ylabel style=blue, axis y line*=left, symbolic x coords={2006, 2007, 2008, 2009, 2010, 2011, 2012, 2013, 2014}, ] \addplot+[ybar, blue, bar shift=-0.2cm] coordinates { (2006, 2.29e-09) (2007, 1.35e-07) (2008, 4.00e-12) (2009, 0.00e+00) (2010, 0.00e+00) (2011, 1.36e-07) (2012, 8.96e-07) (2013, 6.72e-11) (2014, 4.70e-06) }; \end{semilogyaxis} \begin{axis}[ ybar, width = 0.9\linewidth, ymin=0, ymax=100, ylabel = {Anteil an Gesamtereignissen (\%)}, yticklabel style=red, ytick style=red, ylabel style=red, axis y line*=right, symbolic x coords={2006, 2007, 2008, 2009, 2010, 2011, 2012, 2013, 2014}, % hide x axis ] \addplot+[ybar, red, bar shift=0.2cm] coordinates { (2006, 2.36) (2007, 42.11) (2008, 0.00) (2009, 0.00) (2010, 0.00) (2011, 40.37) (2012, 57.07) (2013, 0.05) (2014, 106.03) }; \end{axis} \end{tikzpicture} \end{document} Die beiden x-Achsen liegen nicht übereinander.
|
|
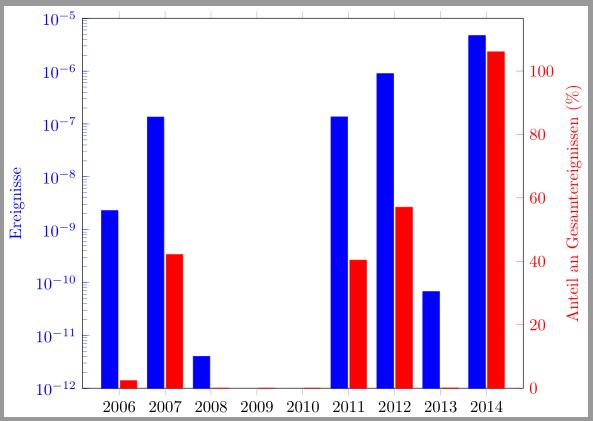
In Deinem zweiten Plot ist der letzte Funktionswert größer als Du muss also eigentlich nur
Außerdem würde ich die Sachen, die für beide Plots gleich sind oder sogar sein müssen, an nur einer Stelle einstellen. \documentclass{standalone} \usepackage{pgfplots} \pgfplotsset{compat=newest} \begin{document} \begin{tikzpicture} \pgfplotsset{ every axis/.style={ ybar, width=.9\linewidth, symbolic x coords={2006, 2007, 2008, 2009, 2010, 2011, 2012, 2013, 2014} }} \begin{semilogyaxis}[ log origin=infty, ymin=1.0e-12, ymax=1.0e-05, ylabel={Ereignisse}, yticklabel style=blue, ytick style=blue, ylabel style=blue, axis y line*=left, ] \addplot+[blue, bar shift=-0.2cm] coordinates { (2006, 2.29e-09) (2007, 1.35e-07) (2008, 4.00e-12) (2009, 0.00e+00) (2010, 0.00e+00) (2011, 1.36e-07) (2012, 8.96e-07) (2013, 6.72e-11) (2014, 4.70e-06) }; \end{semilogyaxis} \begin{axis}[ ymin=0, %ymax=107,% <- ymax=100 war zu klein ylabel = {Anteil an Gesamtereignissen (\%)}, yticklabel style=red, ytick style=red, ylabel style=red, axis y line*=right, %hide x axis ] \addplot+[red, bar shift=0.2cm] coordinates { (2006, 2.36) (2007, 42.11) (2008, 0.00) (2009, 0.00) (2010, 0.00) (2011, 40.37) (2012, 57.07) (2013, 0.05) (2014, 106.03) }; \end{axis} \end{tikzpicture} \end{document} |