|
Hallo zusammen, ich bräuchte bitte noch einmal eure Unterstützung. Ich möchte eine DNA-Sequenz erstellen. Ich habe auch schon eine Möglichkeit im I-Net gefunden. Jedoch bin ich nicht so fit in Latex, um die Möglichkeit an meine Bedürfnisse anzupassen. Hier der ursprüngliche Code: \usepackage{xparse} \usepackage{xcolor,booktabs} \ExplSyntaxOn \NewDocumentCommand{\duplexsequence}{mO{}mO{}m} { \mbox { \renewcommand{\arraystretch}{0} \jpaul_ds_duplexsequence:nnnnn { #1 } { #2 } { #3 } { #4 } { #5 } } } \cs_new_protected:Nn \jpaul_ds_duplexsequence:nnnnn {% #1 is the number on the left % #2 is the optional prefix % #3 is the number on the right % #4 is the optional postfix % #5 is the top sequence \jpaul_ds_left:nnn { #1 } { #2 } { #3 } \tl_map_function:nN { #5 } \jpaul_ds_pair:n \jpaul_ds_right:nnn { #3 } { #4 } { #1 } } \cs_new_protected:Nn \jpaul_ds_pair:n { \begin{tabular}{@{}c@{}} \jpaul_ds_top:n { #1 } \\\addlinespace[.6ex] $|$ \\\addlinespace[.6ex] \jpaul_ds_bottom:n { #1 } \end{tabular} } \cs_new_protected:Nn \jpaul_ds_top:n { \textcolor{top}{#1} } \cs_new_protected:Nn \jpaul_ds_bottom:n { \textcolor{bottom} { \str_case:nn { #1 } { {T}{A} {A}{T} {C}{G} {G}{C} } } } \cs_new_protected:Nn \jpaul_ds_left:nnn { \begin{tabular}{@{}r@{}} \smash{\textcolor{top}{#1\quad#2}}\vphantom{T}\\\addlinespace[.6ex] \phantom{$|$}\\\addlinespace[.6ex] \smash{\textcolor{bottom}{#3\quad}}\vphantom{T} \end{tabular} } \cs_new_protected:Nn \jpaul_ds_right:nnn { \begin{tabular}{@{}l@{}} \smash{\textcolor{top}{#2\quad#1}}\vphantom{T}\\\addlinespace[.6ex] \phantom{$|$}\\\addlinespace[.6ex] \smash{\textcolor{bottom}{\quad#3}}\vphantom{T} \end{tabular} } \ExplSyntaxOff \definecolor{top}{RGB}{0,0,0} \definecolor{bottom}{RGB}{0,102,0} \begin{document} \duplexsequence{3$'$}{5$'$}{GGCCTATATCGCTTAACCGCGCACATGGA} \end{document} Daraus resultiert folgendes Ergebnis: Das ist auch völlig in Ordnung und ich brauche es auch so (1. Bild). Aber darüber hinaus brauche ich das ganze auch so, also mit der komplementären RNA und der Möglichkeit am Ende das 3' und 5' zu kommentieren wie auf dem 2. Bild. Außerdem brauche ich noch die Möglichkeit 3er Gruppen zu erstellen und den komplementären Strang selber zu erstellen ohne das 3' und 5' Ende oder bei Wunsch mit, so dass es parallel zum Hauptstrang ist (3. Bild). Es muss auch nicht mit dem genannten Code sein. Bin für jede Alternative offen. Vielen Dank und viele Grüße Suku P.s.: Meine Bilder sind in der Vorschau immer zu groß. Kann ich diese im Vorfeld skalieren, oder passiert das automatisch?
Ergebnis 5 von 6
show 1 more comments
|
|
Hallo zusammen, ich habe mich jetzt gegen texshade entschieden, da mir der Aufwand einfach zu groß war. Mein 1. Problem habe ich doch noch durch die Anpassung des geposteten Codes gelöst. Und die Übersetzung der RNA-Sequenz in die Aminosäuren nehme ich manuell in Angriff, da es keine extrem langen Sequenzen sind und packe das ganze dann in eine Tabelle (tabularx). Hier der Code zu meiner Anpassung des oben genannten Codes: \ExplSyntaxOn \NewDocumentCommand{\duplexsequencerna}{mO{}mO{}m} { \mbox { \renewcommand{\arraystretch}{0} \jpaul_ds_duplexsequencerna:nnnnn { #1 } { #2 } { #3 } { #4 } { #5 } } } \cs_new_protected:Nn \jpaul_ds_duplexsequencerna:nnnnn {% #1 is the number on the left % #2 is the optional prefix % #3 is the number on the right % #4 is the optional postfix % #5 is the top sequence \jpaul_ds_Left:nnn { #1 } { #2 } { #3 } \tl_map_function:nN { #5 } \jpaul_ds_Pair:n \jpaul_ds_Right:nnn { #3 } { #4 } { #1 } } \cs_new_protected:Nn \jpaul_ds_Pair:n { \begin{tabular}{@{}c@{}} \jpaul_ds_Top:n { #1 } \\\addlinespace[.6ex] $|$ \\\addlinespace[.6ex] \jpaul_ds_Bottom:n { #1 } \end{tabular} } \cs_new_protected:Nn \jpaul_ds_Top:n { \textcolor{top}{#1} } \cs_new_protected:Nn \jpaul_ds_Bottom:n { \textcolor{bottom} { \str_case:nn { #1 } { {T}{A} {A}{U} {C}{G} {G}{C} } } } \cs_new_protected:Nn \jpaul_ds_Left:nnn { \begin{tabular}{@{}r@{}} \smash{\textcolor{top}{#1\quad#2}}\vphantom{T}\\\addlinespace[.6ex] \phantom{$|$}\\\addlinespace[.6ex] \smash{\textcolor{bottom}{#3\quad}}\vphantom{T} \end{tabular} } \cs_new_protected:Nn \jpaul_ds_Right:nnn { \begin{tabular}{@{}l@{}} \smash{\textcolor{top}{#2\quad#1\quad DNA-Sequenz}}\vphantom{T}\\\addlinespace[.6ex] \phantom{$|$}\\\addlinespace[.6ex] \smash{\textcolor{bottom}{\quad#3\quad RNA-Sequenz}}\vphantom{T} \end{tabular} } \ExplSyntaxOff Liebe Grüße Suku |

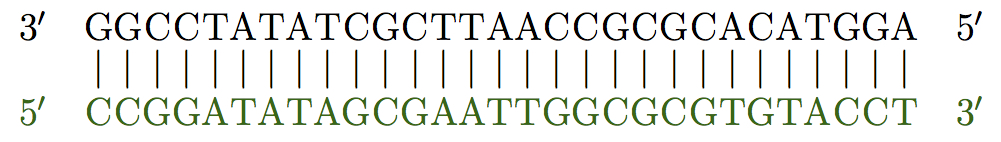


Guten Morgen,
ich habe mich weiter umgeschaut, und das Paket texshade entdeckt. Das Paket scheint meine Wünsche erfüllen zu können. Aber ich bekomme es leider nicht eingebunden. Also usepackage{texshade} alleine scheint nicht zu reichen und mehr Erfahrungen habe ich mit Latex noch nicht. Könnt ihr mir da weiterhelfen? Mir fehlen in der Paket Beschreibung leider auch die Bilder, um mir die gewollten Rosinen heraus zu picken... seufz
Mir würde es erst einmal ausreichen, wenn ich das Paket zum Laufen bekomme, dann kann ich immer noch selber herum basteln.
Vielen Dank und viele Grüße Suku
Ich weiß nicht, wo du ein Problem mit texshade hast. Ich musste nur die Beispieldaten aus dem doc-Ordner kopieren und dann gingen die (bebilderten!) Beispiele aus der Doku sofort.
Hi,
was meinst du denn mit doc-Ordner? Mir ist der Ausdruck noch nicht begegnet.
Viele Grüße Suku
@Suku wenn Du z:b TeX live installiert hast, dann legt TeX live alle Dokumentation (doc) zu den verschiedenen Paketen in einem Ordner ab. Bei mir ist das zB
/usr/local/texlive/2017/texmf-dist/doc/. Die Doku zutexshadeist dann in/usr/local/texlive/2017/texmf-dist/doc/latex/texshade/. Dort finden sich eine ganze Reihe von Beispieldateien@Clemens Vielen Dank für deine Erklärung. Hätte ich das vorher mal gewusst, hätte ich mir zu so einigen Paketen die Zeit fürs googeln sparen können.
Vielleicht auch interessant: http://texwelt.de/wissen/fragen/6725/wo-bzw-wie-finde-ich-die-anleitung-zu-einem-paket-oder-einer-klasse