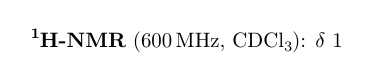|
Ich schreibe gerade an einem Laborbericht, in welchem ich spektroskopische NMR Daten angeben muss - dafür möchte ich das Paket \documentclass{article} \usepackage[utf8]{inputenc} \usepackage{chemmacros} \usechemmodule{all} \begin{document} \begin{experimental}[format=\bfseries] \NMR(600)[CDCl3] \val{1} \end{experimental} \end{document} sieht der Output in etwa so aus: 1H-NMR (600 MHz, CDCl3): ... Ist es möglich, dass ausschließlich das 1H-NMR fett ausgegeben wird, der Rest hingegen normal: 1H-NMR (600 MHz, CDCl3): ...? |
|
Kurze Antwort: nein. Längere Antwort: nicht, ohne eine Interne Funktion zu erweitern. \documentclass{article} \usepackage[utf8]{inputenc} \usepackage[T1]{fontenc} \usepackage{chemmacros} \usechemmodule{spectroscopy} \ExplSyntaxOn \tl_new:N \l__chemmacros_nmr_base_format_tl \chemmacros_define_keys:nn {spectroscopy} { nmr-base-format .tl_set:N = \l__chemmacros_nmr_base_format_tl } \cs_set_protected:Npn \__chemmacros_nmr_base:nn #1#2 { \group_begin: \tl_use:N \l__chemmacros_nmr_base_format_tl \tl_if_blank:VF \g__chemmacros_nmr_element_coupled_tl { \tl_put_left:Nn \g__chemmacros_nmr_element_coupled_tl { \{ } \tl_put_right:Nn \g__chemmacros_nmr_element_coupled_tl { \} } } \tl_put_left:Nn \g__chemmacros_nmr_element_coupled_tl {#2} \chemmacros_chemformula:n { ^{#1} } \bool_if:NTF \l__chemmacros_nmr_parse_bool { \chemformula_ch:nV {} \g__chemmacros_nmr_element_coupled_tl } { \chemmacros_chemformula:V \g__chemmacros_nmr_element_coupled_tl } \tl_use:N \l__chemmacros_nmr_element_method_connector_tl \tl_use:N \l__chemmacros_nmr_method_tl \group_end: } \ExplSyntaxOff \begin{document} \begin{experimental}[nmr-base-format=\bfseries] \NMR(600)[CDCl3] \val{1} \end{experimental} \end{document}
Da die Lösung auf internen Funktionen beruht, wird sie evtl nicht dauerhaft zuverlässig funktionieren. Wenn ich's nicht vergesse, werde ich das offiziell einbauen und die Antwort dann überarbeiten. Anmerkung:Ohne Hack aber mit
Was evtl auch eine Lösung sein könnte. Super! Vielen, vielen Dank, genau das, wonach ich gesucht habe!
(27 Jan '17, 01:29)
maxim87
Hallo, ich stehe im Moment vor dem selben Problem. Leider funktioniert bei mir der "Hack" nicht und ich erhalte folgende Fehlermeldung. Hat jemand eine Idee, wie dies zu beheben ist? Beste Grüße!
(23 Jan '20, 14:01)
barmaritter
|