|
Beim compilieren des Codes unten erhielt ich die Fehlermeldung:
Was mache ich falsch ? \documentclass[draft]{scrbook} \usepackage{ngerman} \usepackage[utf8]{inputenc} \usepackage{tikz,chemfig} \usetikzlibrary{shadows,arrows,intersections} \begin{document} \tikzset{ obrace/.style={left delimiter={[},inner sep=3pt}, cbrace/.style={right delimiter={]},inner sep=3pt}, braces/.style={left delimiter={[},right delimiter={]},inner sep=3pt} } \setatomsep{2em} \schemestart \chemfig{H-C(=[:-60]O)-[:60]O-H} \arrow(--M1[obrace]){-U>[\scriptsize\chemfig{H_2SO_4^{}}][\scriptsize\chemfig{HSO_4^\ominus}][][.25]}% [,1.5,shorten >=6pt] \chemfig{H-@{a2}C(-[:60]O-H)(-[:30,.5,,,draw=none]{\scriptstyle\oplus})-[:-60]O-H} \arrow(--[cbrace]){<->} \schemestop \end{document} |
|
Du musst für
|

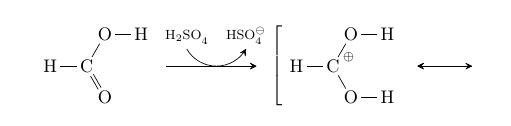
Du musst für
left delimiterundright delimiterwenn ich mich recht erinnere die TikZ-librarymatrixladen.Danke ja das war der Grund.
Sagmal gibt es eine Möglichkeit herauszubekommen welche tikzlibraries ein code benötigt, dem man nicht so ganz versteht, damit solche Fehler zukünftig unterbleiben. ?
@butts das steht ausführlich im pgfmanual und in diesem speziellen Fall steht es auch im
chemfig-Handbuch, wie ich gerade noch mal überprüft habe, und zwar im Abschnitt wenn\subschemebesprochen wird.